|
|
iPS細胞樹立受託サービス(RNAリプログラミング)
|
|
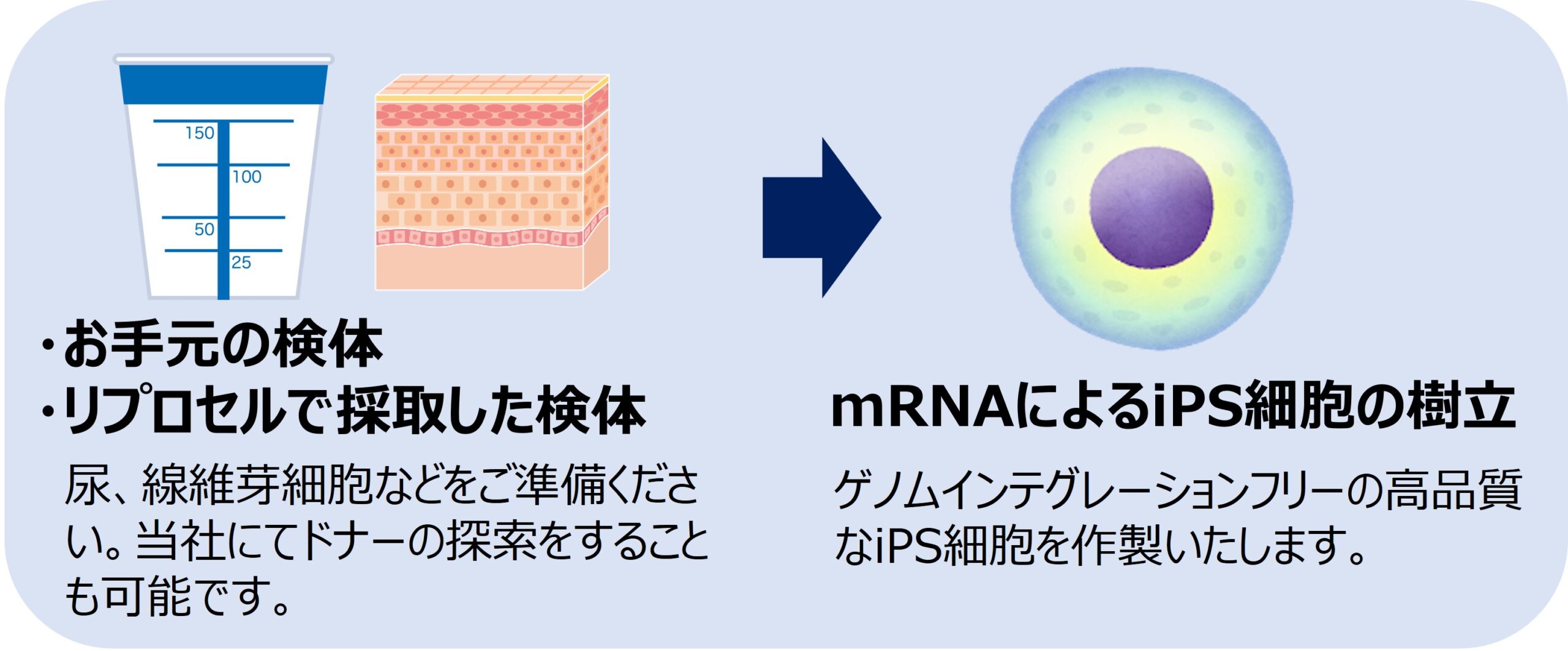
当社では、導入因子のゲノムへのインテグレーションが起こらず安全性が高いことが知られているRNA法を用いてiPS細胞を樹立いたします。 患者様の尿、線維芽細胞をお送りいただければ、お手元の患者様由来のiPS細胞が手に入ります。また、リプロセルグループにて、ご要望の患者様から検体を採取することが可能であり、研究用途に合わせた多様な疾患患者由来のiPS細胞を入手することができます。 加えて当社では、ゲノム編集サービスや目的の細胞への分化誘導サービスも承っていますので、ワンストップで、必要な細胞をお届けできます。
樹立サービス概要
|
品番 |
納品物 |
検定項目 |
納期・価格 |
|---|---|---|---|
| RCSE001 | 3クローン 3バイアル(各クローン) |
・細胞の形態
・細胞の増殖
・未分化マーカーの発現(免疫染色)<v/p>
|
お問い合わせ下さい |
RNAリプログラミングキットの使用報告
RNAリプログラミング特徴と優位性
(1) iPS細胞の樹立(RNAリプログラミング)
iPS細胞は2006年にマウスで、2007年にヒトで初めて樹立された(1、2、3、4)。当時は4つの因子(Oct4、Sox2、Klf4、c-Myc; OSKM)をレトロウイルスベクターを用いて線維芽細胞に遺伝子導入し初期化を誘導していたが、iPS細胞の樹立方法(リプログラミング方法)は改良され続けている。 特に、外来遺伝子の再活性化により腫瘍が発生するリスクが議論されて以来(5)、レトロウイルスベクターやレンチウイルスベクターなど導入因子を染色体に組み込むウイルスベクターの使用が敬遠されてきた。これらの点を踏まえて、iPS細胞を安全性の観点から評価すると、1)導入因子の残存性が小さいこと2)染色体の安定性が高いことが必要となる(6)。加えて、安定的な製造が必要となる産業化のの観点からは、3)クローン間差が小さいことも重要である。実際の再生医療用途で使用されたiPS細胞は複数のクローンの中から多くの選別を経て、臨床試験に使用可能なクローンが選ばれた(7)。 現在使用されている樹立方法を比較すると、エピゾーマルベクターでは、導入因子としてDNAを用いることから染色体への取り込みの可能性を完全に否定することは難しいと報告されている(8)。また、センダイウイルスベクターを用いたiPS細胞の樹立においては、ゲノムへの取り込みの危険性は回避されているが、導入因子の長期残存性が報告されている(9)。 このような状況の中で、2010年にCell Stem Cell誌にてRNAを用いたリプログラミング方法が報告された(10、11)。RNAリプログラミングでは、導入因子をRNAとして細胞に導入し、細胞質に留まったRNAから各種の初期化因子が翻訳されて細胞が初期化される。さらに、RNAは細胞内で速やかに分解される(図1)ことから、導入因子の残存性を気にすることなく、染色体の安定性が高く、クローン間差が小さいRNA-iPS細胞は、今後の再生医療利用および産業利用に最適なiPS細胞である。
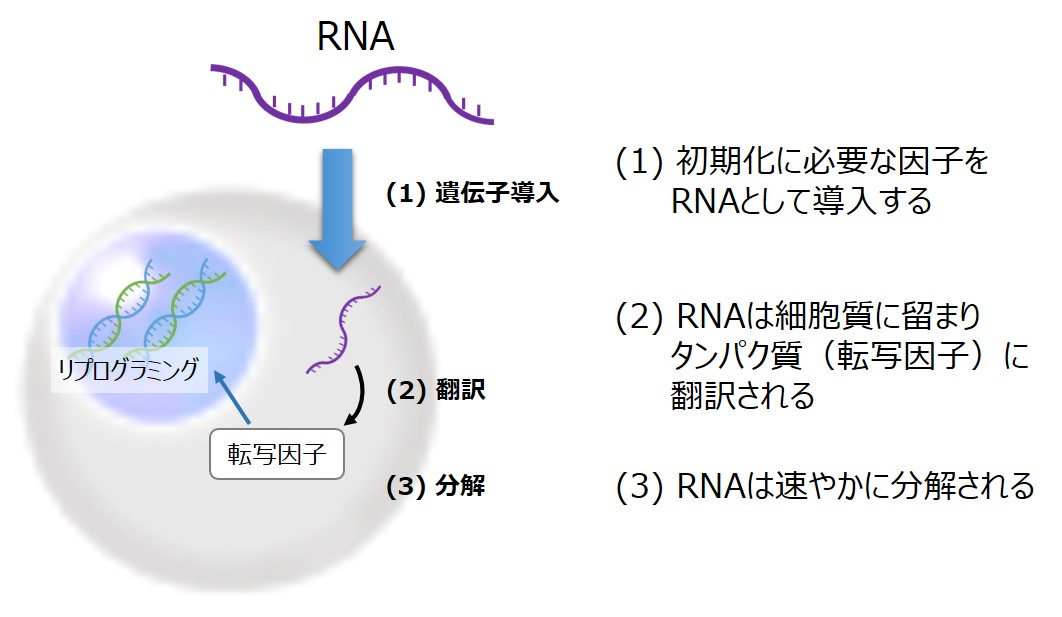
図1.RNAによるiPS細胞の樹立
(2) RNAリプログラミング法の変遷
RNAリプログラミング法は、2012年に発売を開始した第一世代の製品から、2015年には第二世代に、そして2016年には第三世代の技術へと進歩してきている(図2)。

図2.RNAリプログラミング法の変遷
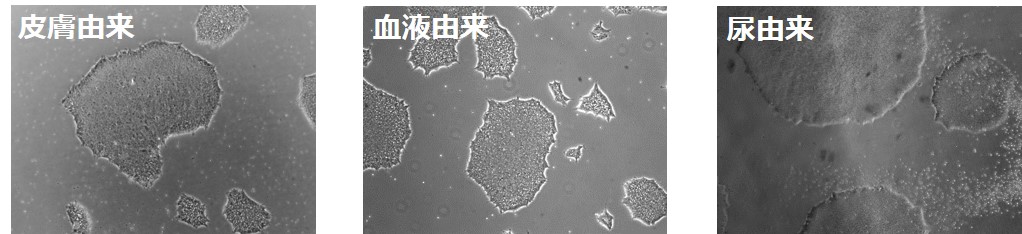
図3.皮膚、血液、尿から樹立されたiPS細胞
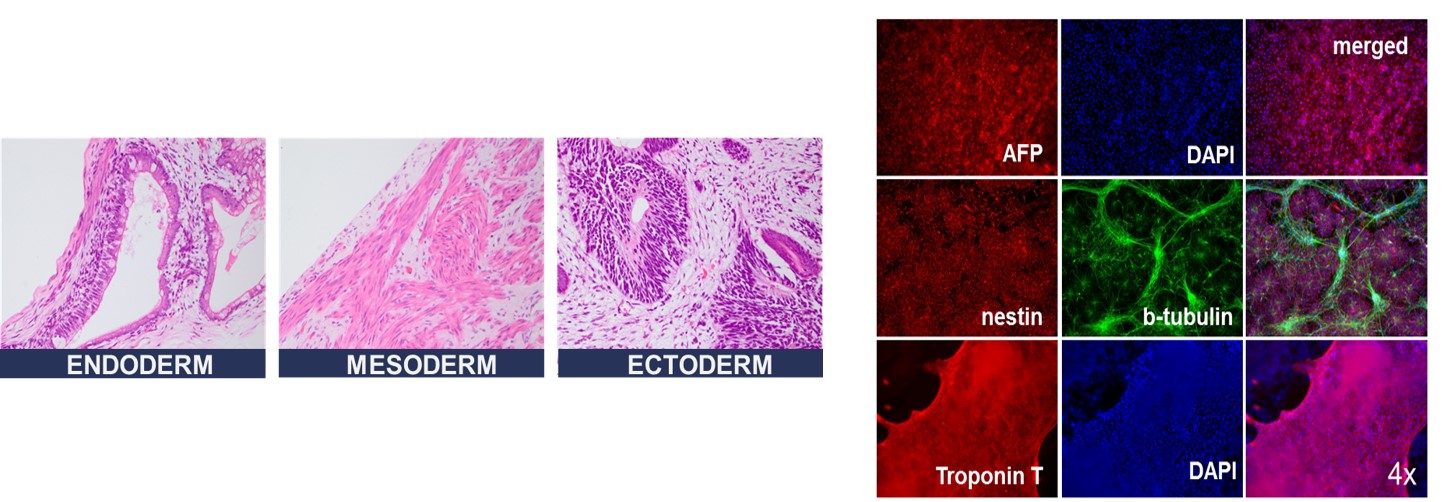
図4.三胚葉への分化能(左:テラトーマ形成、右:in vitro 分化)
(3)造血前駆細胞への分化誘導
今後の再生医療において、iPS細胞から分化誘導される血球細胞に大きな期待が寄せられています。様々な由来のiPS細胞から血球系への細胞への分化誘導能を確認することを目的として、血球細胞由来細胞ではない異なる2株から造血前駆細胞への分化誘導を試みました。RNA法を用いて樹立した血液中の血管内皮前駆細胞(Endothelial Progenitor Cells: EPC)由来のiPS細胞(RPChiPS7713G1)、同様にRNA法を用いて樹立した線維芽細胞由来のiPS細胞(B3-3A)を使用しました。由来の細胞種によらず、誘導開始12日後に造血幹細胞様の綺麗な球形を保った細胞を誘導する事に成功しました(図5)。
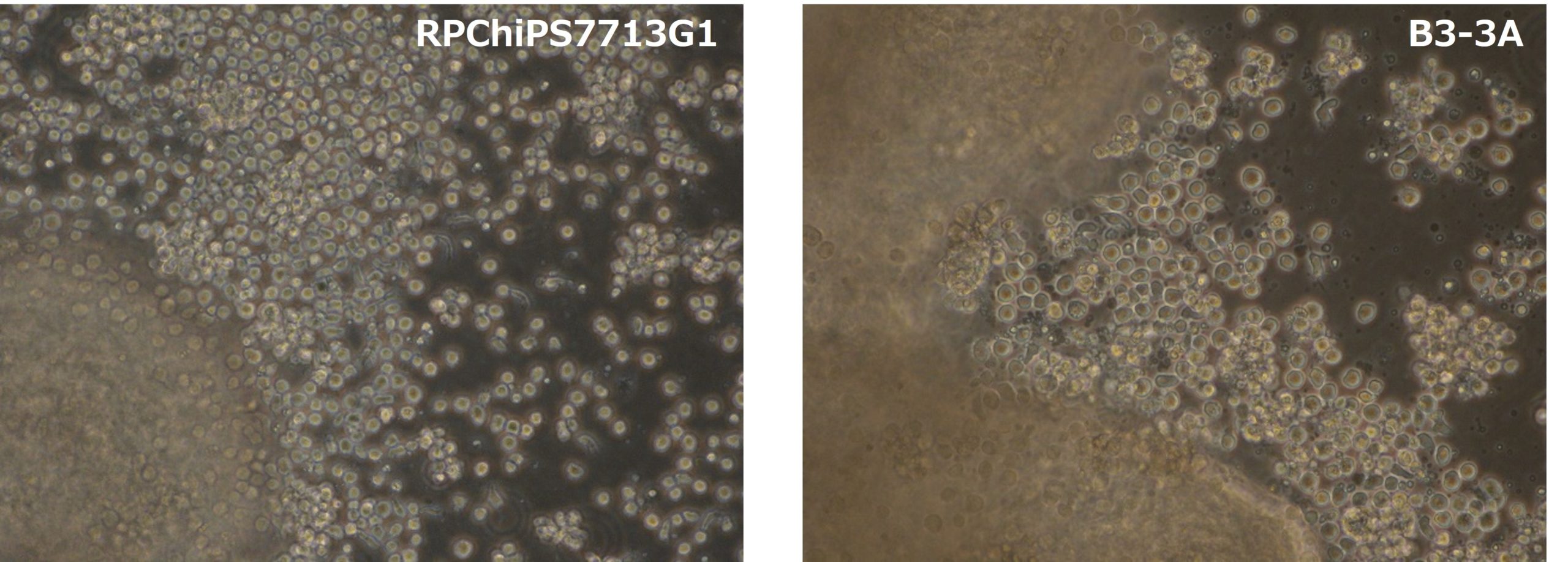
図5.iPS細胞から分化誘導された造血前駆細胞(誘導開始後12日)
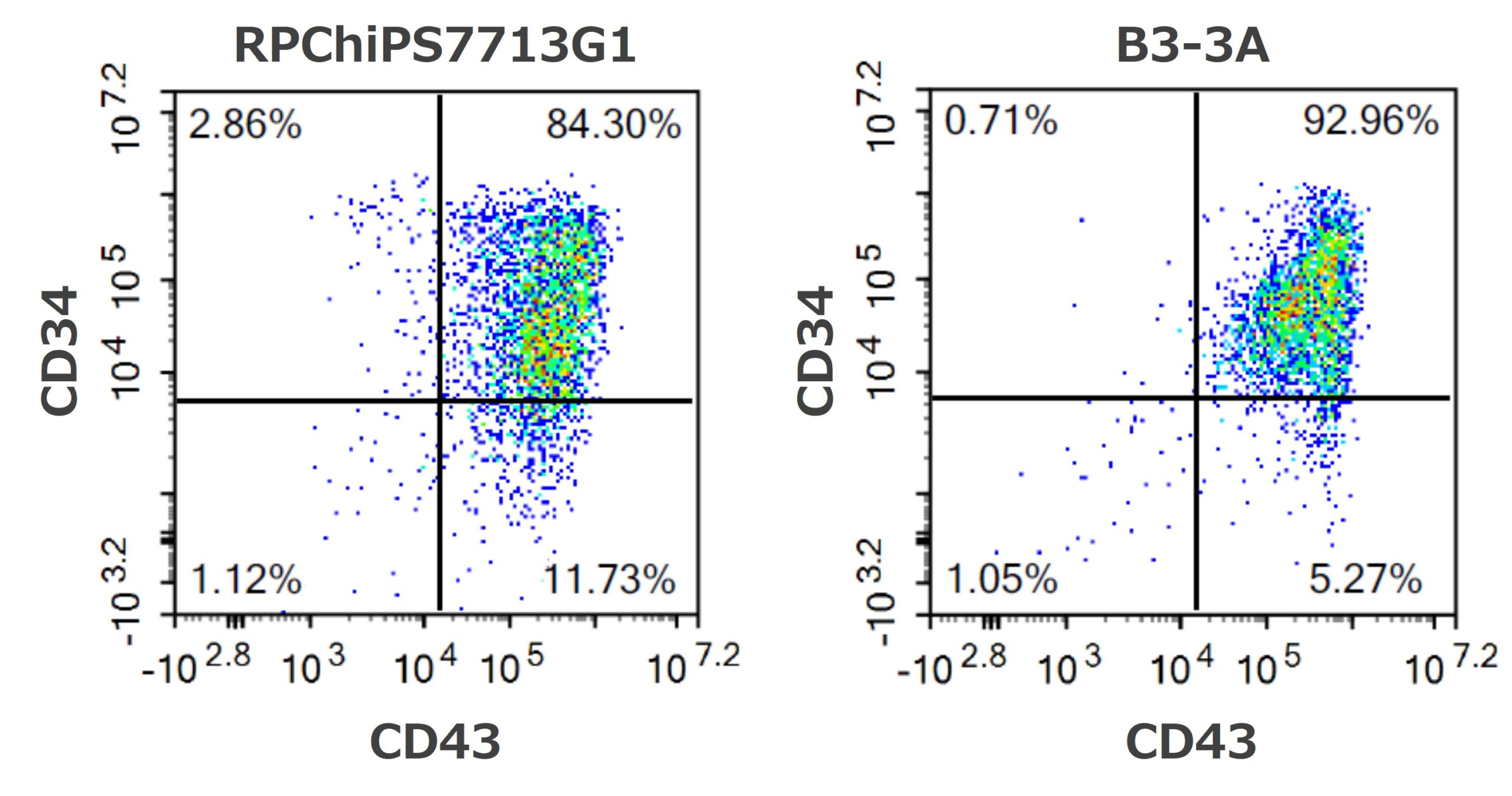
図6.造血前駆細胞マーカー(CD34陽性・CD43陽性細胞)の発現解析(誘導開始後12日)
(4) RNAリプログラミング法で樹立されたiPS細胞の優位性
・導入因子の残存性が低い RNA-iPS細胞は、導入因子にRNAを用いており、RNAは細胞内で速やかに分解されることからiPS細胞内での残存性を気にすることなく使用可能である。iPS細胞を樹立後、2~3継代培養したiPS細胞では、導入されたRNAは検出限界以下になっていた(図7)。

図7.導入されたRNAの消失
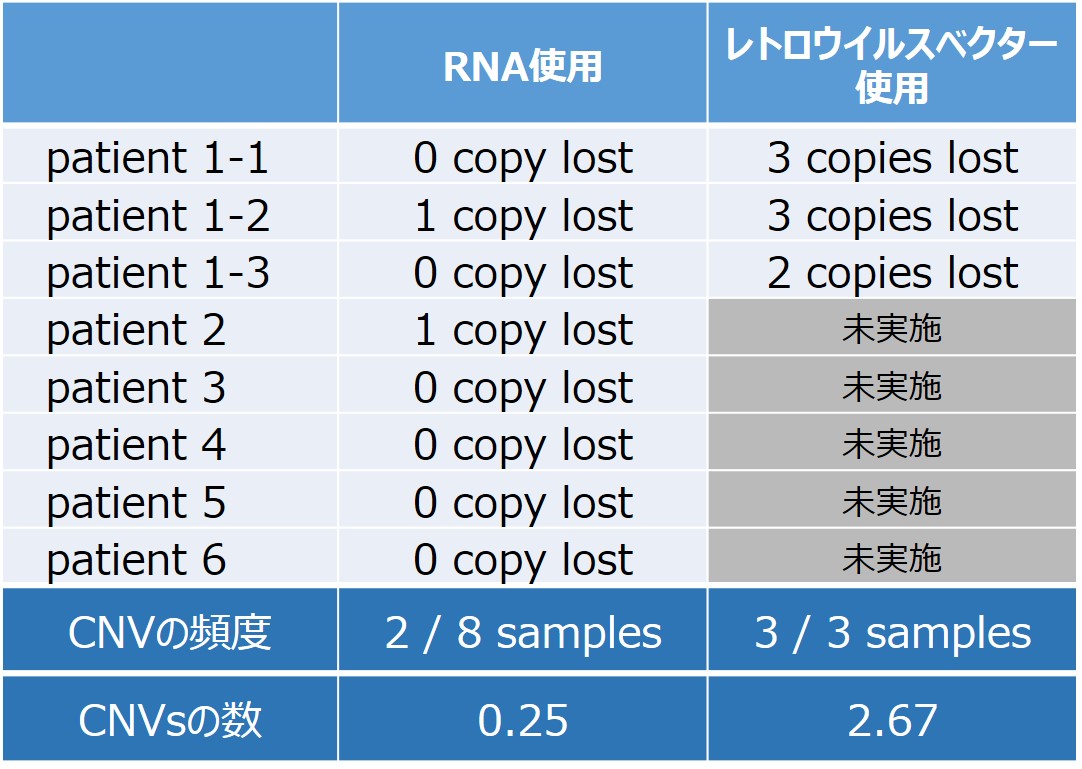
表1.RNAで樹立されたiPS細胞のCNV

図8.クローン間差が小さいiPS細胞
(5) 最後に
ヒトiPS細胞が報告されてから10年以上が経過し樹立方法に関して、より良い方法が開発されてきた。特に、RNAリプログラミング法は、当初より優れた方法であると期待されており、現在は第三世代まで発展してきている。iPS細胞の本格的な商業利用や再生医療への応用が期待されている中で、使用されるiPS細胞の重要となる特徴が定められつつある。まず、注目すべきポイントは安全性(導入因子の残存性および染色体の安定性)である。これまでに述べてきたようにRNA-iPS細胞は、この点に関して大きな優位性を有している。加えて、商業化において重要なポイントとなる製造および最終製品の安定供給においても、RNA-iPS細胞を使用するメリットは大きい。RNA-iPS細胞は尿からの樹立が可能となったこともあり、今後の使用範囲が大きく広がることが期待されている。
参考文献
-
Takahashi K, Yamanaka S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 2006 126(4):663-76.
-
Wernig M, Meissner A, Foreman R, Brambrink T, Ku M, Hochedlinger K, Bernstein B.E, Jaenisch R. In vitro reprogramming of fibroblasts into a pluripotent ES-cell-like state. Nature. 2007 448(7151):318-24.
-
Takhashi K, Tanabe K, Ohnuki M, Narita M, Ichisaka T, Tomoda K, Yamanaka S. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 2007 131(5):861-72.
-
Yu, J, Vodyanik M.A, Smuga-Otto K, Antosiewicz-Bourget J, Frane J.L, Tian S, Nie J, Jonsdottir G.A, Ruotti V, Stewart R, Slukvin I.I, Thomson J.A. Induced pluripotent stem cell lines derived from human somatic cells. Science, 2007 318(5858):1917-20.
-
Okita K, Ichisaka T, Yamanaka S. Generation of germline-competent induced pluripotent stem cells. Nature. 2007 Jul 19;448(7151):313-7.
-
厚生労働省医政局研究開発振興課 「特定認定再生医療等委員会におけるヒト多能性幹細胞を用いる再生医療等提供計画の造腫瘍性評価の審査のポイント」2016
-
Mandai M, Watanabe A, Kurimoto Y, Hirami Y, Morinaga C, Daimon T, Fujihara M, Akimaru H, Sakai N, Shibata Y, Terada M, Nomiya Y, Tanishima S, Nakamura M, Kamao H, Sugita S, Onishi A, Ito T, Fujita K, Kawamata S, Go MJ, Shinohara C, Hata KI, Sawada M, Yamamoto M, Ohta S, Ohara Y, Yoshida K, Kuwahara J, Kitano Y, Amano N, Umekage M, Kitaoka F, Tanaka A, Okada C, Takasu N, Ogawa S, Yamanaka S, Takahashi M. Autologous Induced Stem-Cell-Derived Retinal Cells for Macular Degeneration. N Engl J Med. 2017 Mar 16;376(11):1038-1046.
-
Okita K, Matsumura Y, Sato Y, Okada A, Morizane A, Okamoto S, Hong H, Nakagawa M, Tanabe K, Tezuka K, Shibata T, Kunisada T, Takahashi M, Takahashi J, Saji H, Yamanaka S. A more efficient method to generate integration-free human iPS cells. Nat Methods. 2011 May;8(5):409-12.
-
Agu CA, Soares FA, Alderton A, Patel M, Ansari R, Patel S, Forrest S, Yang F, Lineham J, Vallier L, Kirton CM. Successful Generation of Human Induced Pluripotent Stem Cell Lines from Blood Samples Held at Room Temperature for up to 48 hr. Stem Cell Reports. 20115 Oct 13;5(4):660-71.
-
Warren, L., Manos, P.D., Ahfeldt, T., Loh, Y.H., Li, H., Lau, F., Ebina, W., Mandal, P.K., Smith, Z.D., Meissner, A., Daley, D.Q., Brack, A.S., Collins, J.J., Cowan, C., Schlaeger, T.M., Rossi, D.J. Highly Efficient Reprogramming to Pluripotency and Directed Differentiation of Human Cells with Synthetic Modified mRNA. Cell Stem Cell. 2010 7:618.
-
Yakubov, E., Rechavi, G., Rozenblatt, S., Givol, D. Reprogramming of Human Fibroblasts to Pluripotent Stem Cells using mRNA of Four Transcription Factors. Biochem Biophys Res Commun. 2010 394:189.
-
Sridhar A, Ohlemacher SK, Langer KB, Meyer JS. Robust Differentiation of mRNA-Reprogrammed Human Induced Pluripotent Stem Cells Toward a Retinal Lineage. Stem Cells Transl Med. 2016 Apr;5(4):417-26.
-
Schlaeger TM, Daheron L, Brickler TR, Entwisle S, Chan K, Cianci A, DeVine A, Ettenger A, Fitzgerald K, Godfrey M, Gupta D, McPherson J, Malwadkar P, Gupta M, Bell B, Doi A, Jung N, Li X, Lynes MS, Brookes E, Cherry AB, Demirbas D, Tsankov AM, Zon LI, Rubin LL, Feinberg AP, Meissner A, Cowan CA, Daley GQ. A comparison of non-integrating reprogramming methods. Nat Biotechnol. 2015 Jan;33(1):58-63.
-
Paull D, Sevilla A, Zhou H, Hahn AK, Kim H, Napolitano C, Tsankov A, Shang L, Krumholz K, Jagadeesan P, Woodard CM, Sun B, Vilboux T, Zimmer M, Forero E, Moroziewicz DN, Martinez H, Malicdan MC, Weiss KA, Vensand LB, Dusenberry CR, Polus H, Sy KT, Kahler DJ, Gahl WA, Solomon SL, Chang S, Meissner A, Eggan K, Noggle SA. Automated, high-throughput derivation, characterization and differentiation of induced pluripotent stem cells. Nat Methods. 2015 Sep;12(9):885-92.
